| Investigador Principal (IP): Carracedo Álvarez, Angel | Código: IPT-2011-0963-900000 | ||
| Convocatoria: ayudas Acción Estratégica en Salud 2012 (AES 2012) – ISCIII | |||
| Presupuesto: 940.986,00€ | Fecha inicio: dd/mm/2011 | Duración: 3 años | |
| Objeto del proyecto:
Al plantear este estudio, se perseguía aunar la experiencia de un socio- promotor industrial, Integromics, y otro de carácter clínico asistencial y público, la FPGMX, parael desarrollo de la primera plataforma de análisis e interpretación clínica de datos de ultra-secuenciación en España.La ejecución de este proyecto permitiría que el análisis genómico personalizado sea la cuarta tecnología de aplicación para la investigación biomédica. Esto aplicado en y para un entorno de asistencia sanitaria ordinaria, paliaría/ aliviaría sin duda laactual falta de aplicaciones necesarias, así como ayudaría a disminuírla cargade trabajo de los facultativos. Palabras clave:plataforma de análisis, análisis genómico personalizado, ultrasecuenciación masiva (NGS), tecnología, aplicación, innovación, medicina genómica personalizada, clínica. |
|||
EQUIPO
| Apellidos | Nombre | Titulación/ Categoría | Entidad |
| González Couto | Eduardo | Dir. Técnico I+D+i/ coordinador | Integromics |
| Foissac | Sylvain | Bioinformatica, doctor/ científico | |
| Proença | Henrique | Dir. Desarrollo, ingeniero/ colaborador | |
| Panadero | Joaquín | Informática, doctor/ científico | |
| Santos | Luis | Informática, doctor/ asistencia técnica | |
| Cuesta | Isabel | ¿?, doctora/ soporte científico | |
| Varvanov Soft. | — | Empresa software/ subcontrata | |
| Carracedo Álvarez | Angel | Profesor de Medicina Legal y
Directorde la FPGMX / científico |
USC y FPGMX |
| Barros Angueira | Francisco | ¿?, doctor/ científico | CEGEN, FPGMX |
| Sobrino Rey | Beatriz | ¿?, doctora/ científica | FPGMX |
| Amigo Lechuga | Jorge | ¿?, doctor/ científico | FPGMX |
IP: Investigador Principal
ENTIDADES PARTICIPANTES Y COLABORADORAS
| Nombre | Siglas | Tipo/ Categoría |
| Centro Nacional de Genotipado | CEGEN | Centro público de investigación |
| Fundación Pública Galega de Medicina Xenómica | FPGMX | Organismo público |
| Complexo Hospitalario Universitario de Santiago | CHUS | Hospital público |
| Complexo Hospitalario Universitario de Vigo | CHUVI | Hospital público |
| Integromics S.A. | ¿? | Empresa privada |
| Servizo Galego de Saúde | SERGAS | Organismo público |
| Universidade de Santiago de Compostela | USC | Organismo público |
Antecedentes/ Punto de partida.
La empresa andaluza Integromics S.L. posee unasólida experiencia en el desarrollo, comercialización y aplicaciones de productos bioinformáticos,el campo de las Ciencias de la Vida, y en base al uso de equipos de alto rendimiento para el análisis de los datos obtenidos/ adquiridos/ producidos.
Integromics S.L. propone liderar el proyecto de desarrollo de la primera plataforma de análisis e interpretación clínica de los datos de ultra-secuenciación. El proyecto sebeneficiará/ beneficia de la experiencia del Grupo de Medicina Xenómica en el laboratorio hospitalario de la Fundación Pública Galega de Medicina Xenómica (FPGMX), que se ocupa de más de 12000 casos anuales,utilizando así mismo los datos obtenidos gracias al primer equipo de ultra-secuenciación (o NGS, por sus siglas en inglés). Para ello dicho equipo/ equipamiento se adaptará/ ha adaptado a un uso clínico del tipo “Ion Personal Genome Machine™” (Ion Torrent; (“lowcost”-NGS o LC-NGS ) y que se encuentra instalado en la Fundación.
Las muestras relevantes en el ámbito clínico analizadas han sido/ son/ serán, sangre periférica, tejidos fijados e incluidos en parafina (FFPE) y sangre seca en papel, para las que se han obtenido/ obtendrán las secuencias correspondientescon los chips del Ion Torrent 314, 316 y 318,respectivamente.
Dichas secuencias, también llamadas paneles o colecciones de regiones genómicas y exomas completos, se obtendránsegún las necesidades específicas de las patologías a estudiar.
A continuación se han identificado/ identificarán las mutaciones detectadas en cada paciente, que servirán para generar automáticamente informes/ reportespara uso en clínica, particularmente, para su explotación en medicina genómica personalizada.
Objetivos técnicos específicos.
La solución de análisis e interpretación de los datos de genómica personal requiere combinar la optimización y estandarización de las técnicas de laboratorio con la creación de un conjunto de herramientas computacionales que incluyen (i) la implementación de módulos de software específicos para el control de calidad de las variaciones genómicas identificadas en cada individuo, (ii) su caracterización genómica, (iii) análisis e interpretación, necesitando la integración de fuentes de información sobre asociaciones con patologías, y (iv) la validación de los resultados obtenidos por una técnica de análisis independiente como lo son la PCR cuantitativa (qPCR) o la PCR en tiempo real (RT-qPCR).
Dentro de un marco único y una visión común, el proyecto se abordará desde dos perspectivas, para poder concebir una metodología de trabajo de flujo único pero autónoma para cada socio.
Por una parte la perspectiva del laboratorio clínico con las necesidades de optimización, reproducibilidad, estandarización de los procesos y de disponer de una herramienta de análisis que permita transformar los análisis genómicos brutos (secuencias, variantes, mutaciones) en resultados directamente interpretable para los profesionales de la medicina.
Por otra parte la plataforma computacional desarrollada por la empresa coordinadora (Integromics),como parte de su estrategia corporativa a travésde la nueva línea modular de productos OmicsOffice™. La ejecución de este proyecto permitirá que el análisis genómico personalizado sea la cuarta tecnología de aplicación, siendo los micro-arrays la primera (IntegromicsBiomarker Discovery®), RT-qPCR la segunda (StatMiner®) y la tercera, y última, ultra- secuenciación del ARN (RNA-seq, con la aplicación SeqSolve™). El lanzamiento de OmicsOffice se realizará en el curso del 2011 sobre la plataforma industrial TIBCO Spotfire®, integrando las tres primeras tecnologías mencionadas. Este proyecto permitirá añadir una cuarta tecnología que beneficiará inmediatamente de las combinaciones con las tres otras tecnologías, permitiendo de este modo la validación y el seguimiento integrados, por ejemplo, de mutaciones en el genoma de un paciente por PCR.
Para cubrir el objetivo general, este proyecto cumplirá con los siguientes objetivos:
1) optimización y adaptación de los flujos de laboratorio de procesamiento de las muestras y de preparación de las librerías para un uso compatible con aplicaciones clínicas de la plataforma Ion Torrent
2) optimización y adaptación de los flujos de análisis primario y segundario de los datos de ultra- secuenciación para el uso clínico
3) análisis terciario, control de calidad e interpretación clínica (un caso de uso clínico, inicialmente Retraso Mental en pacientes pediátricos)
4) análisis terciario, control de calidad e interpretación clínica (múltiples casos de uso)
5) generación automática de reportes clínicos
6) producción del software comercial “OmicsOffice™ for Personal Genomics”
El sistema bioinformático de análisis terciario e interpretación de los datos partirá de las listas de variaciones identificadas en el análisis segundario a partir de los datos de ultra secuenciación. Como paso adicional de control de calidad, se efectuará la caracterización genómica y funcional de las variaciones, seguida por el análisis integrativo para poder generar reportes adaptados al enfoque del caso clínico. Este sistema será acopladoa la “suite” OmicsOffice de Integromics para su comercialización, abriendo de esta forma la puerta a la integración de varias tecnologías de alto rendimiento en una sola plataforma clínica para los usuarios.
Publicaciones y estado del arte previos a la realización del proyecto/ estudio. Relevancia.
La creación de una plataforma bioinformática de análisis e interpretación clínica de los datos de ultra- secuenciación, es una absoluta necesidad en el campo de aplicación clínico de la medicina genómica. La demanda biomédica para la tecnología NGS supera la capacidad disponiblehoy en día, sobre todo en relación con la disponibilidad de profesionales de la bioinformática genómica. Lo que propone/ se propone nuestro consorcio es el desarrollo de nuevas herramientas bioinformáticas que permitanla automatización de esta última parte del proceso a corto plazo. Para ello contamos con el bagaje/ la combinación de la experiencia bio-medica y de diagnostico clínico de un laboratorio académico tal como el grupo de Xenómica y la técnica, la capacidad de desarrollo industrial y de comercialización del socio empresarial,la especialista en bioinformáticaIntegromicsS.L..
Una de los organismos de referencia en nuestro ámbito, el “Biotechnology and BiologicalSciencesResearch Council” (BBSRC) inglés,en un estudio nacional sobre ultra-secuenciación (11), señala que el mayor cuello de botella hoy en díaes el desarrollo de herramientas bioinformáticas de uso simple o user-friendly,Dichas herramientas deben permitir la extracción de conocimiento en base a/ guiadas por preguntas biológicas. Según dichos expertos, el vacío decompetencias actual en el análisis e interpretación de los datos requiere de nuevas herramientas de caractermulti-disciplinario y con interfaces gráficas, que simplifiquen el uso de los programas.
El consorcio formado por el grupo Xenómica e Integromics, aúna/ conjuga la participación de expertos y su interdisciplinaridad,con el objetivo/ la misión de crear herramientas de uso simple y que satisfagan las necesidades/ demandas actuales de usuarios científicos o médicos. A medio- largo plazo este consorcio aspira y trabaja/ sentará/ sienta las bases para que la secuenciación del genoma y/ o el exoma se conviertan en rutina en el ámbito clínico; serían aplicables desde la atención primaria hasta en áreas de especialización tales como, por ejemplo, la oncología.
Data generation, Green et al., 2011.
The Lancet, Ashley et al., 2010.
la interpretación del genoma por un millón de dólares” (Davies 2010).
“Mental Retardationdatabase”, http://grenada.lumc.nl/LOVD2/MR/
Genes/ X cromosome (Tarpey et al. 2009).
Integrative Genomics Viewer (www.broadinstitute.org/igv)
dbSNP (ncbi.nlm.nih.gov/snp).
HapMap (www.hapmap.org).
1000Genomes Project (1000genomes.org).
http://www.ingenuity.com/news/html/pr_110405_integromics.html
Consorcio “Human Variome Project” (www.humanvariomeproject.org).
Biotechnology and Biological Sciences Research Council” (BBSRC) (accesolibre: http://www.bbsrc.ac.uk/organisation/policies/reviews/scientific-areas/1102-next-generation-sequencing-review.aspx). Fecha de publicación: 9 de Abril 201X
Mercado(s) diana/ objetivo.
El proyecto va expresamente dirigido al mercado clínico de la medicina genómica (análisis genómico personalizado).
Propiedad intelectual/ protección y derechos sobre los activos generados.
A nivel de productos bioinformáticos desarrollados y comercializados, no existen patentes de software, por lo cual Integromics protege los productos y sus marcar (propiedad intelectual, PI) registrándolos para su comercialización y usos posteriores. Este régimen se ha venido aplicando a todos los productos de la empresa hasta la fecha.
Actualmente existe un consejero dedicado plenamente a la gestión de la PI y dos bufetes internacionales de abogados con sede en los EEUU que le dan soporte.Integromics obtiene así una perspectiva global de la situación de IP con un nivel de exigencia máximo. Ésta estrategia de PI distingue a nuestra empresa de otras españolas que también hacen I+D en este ámbito y la posiciona favorablemente para captar clientes/ interés en el sector farmacéutico, sobre todo en el mercado norteamericano, para el que laPI es una de sus máximas prioridades.
En cuanto al grupo de Medicina Xenómica, cabe destacar que en los últimos 5 años ha desarrollado 9 patentes, varias de ellas extendidas internacionalmente y en explotación.Dichas patentes cubren diversos aspectos de la genética aplicada al diagnóstico y a la medicina forense, destacando las dedicadas a la farmacogenética y al diagnóstico genético. Así mismoel grupo ha dado lugar a2spin- offs: “Health-in-Code”, en colaboración con el Servicio de Cardiología del Hospital Juan Canalejo y “Allelyus” en colaboración con el grupo de Farmacología de la USC.
Impacto. Beneficios esperados en el sector y en la sociedad al inicio del proyecto/ estudio.
El éxito/ cumplimiento de los objetivos del proyecto supone una reducción significativa de los tiempos de procesado de las secuencias obtenidas en plataformas de ultra-secuenciación hasta llegar al paciente y para el personal clínico encargado de su manejo. Se obtiene/ obtendrá una herramienta que permitirá sortear el principal escollo en la actualidad para la aplicación/ uso rutinario de la genómica en la clínica, como son el coste del análisis e información. A la fecha del inicio del proyecto/ estudio(i) había carencia de profesionales con la adecuada capacitación/ formación en bioinformática, (ii) el tiempo de análisis requerido por paciente era de semanas – meses, (iii) no todos los profesionales sanitarios habían sido entrenados para interpretar un exoma o un panel grande de genes.
La disponibilidad de protocolos y herramientas como las propuestas/ a desarrollar/ desarrolladasen este proyecto, seránimprescindibles para el impulso de la genómica asistencial. Su uso generalizadosupondrá cambios radicales en la asistencia al paciente debido precisamente a la progresiva implicacióndel personal clínico en su utilización y dominio de la(s) técnica(s).
Plan de trabajo y cronograma.
Detalle de las etapas en el plan de trabajo se recoge en la tabla 1 a continuación:
| Nº | Actividades y tareas | |
| 1 | Optimización y adaptación de los flujos de laboratorio de procesamiento de las muestras y de preparación de las librerías para un uso compatible con aplicaciones clínicas de la plataforma Ion Torrent | |
| 1.1 | Optimización para uso clínico de protocolos de preparación de librerías de ADN (a partir de muestras FFPE, papel, sangre). | |
| 1.2 | Validación de los resultados obtenidos con los protocolos optimizados. | |
| 1.3 | Difusión de las directrices óptimas para las aplicaciones clínicas. | |
| 2 | Optimización y adaptación de los flujos de análisis primario y secundario de los datos de ultra-secuenciación para eluso clínico | |
| 2.1 | Optimización de la secuenciación (Ion Torrent) para uso clínico. | |
| 2.2 | Selección y optimización de los algoritmos de mapeo de los “short reads”. | |
| 2.3 | Selección y optimización de los algoritmos de “variantcalling”. | |
| 3 | Análisis terciario, control de calidad e interpretación clínica (un caso de uso clínico, inicialmente Retraso Mental enpacientes pediátricos) | |
| 3.1 | Análisis de requerimientos y funcional del módulo de control de calidad y caracterización de las mutacionesidentificadas. | |
| 3.2 | Diseño e implementación del módulo de control de calidad y caracterización de las mutaciones identificadas. | |
| 3.3 | Análisis de requerimientos y funcional del módulo de interpretación clínica para la patología prueba de concepto. | |
| 3.4 | Diseño e implementación del módulo de interpretación clínica para la patología prueba de concepto. | |
| 3.4.1
3.4.2 3.4.3 |
Diseño panel de genes para estudio genético del Retraso Mental y preparación de librerías.
Preparación de muestras de población de pacientes y secuenciación. Evaluación del módulo de interpretación clínica. Validación del sistema. |
|
| 4 | Análisis terciario, control de calidad e interpretación clínica (múltiples casos de uso) | |
| 4.1 | Análisis de requerimientos y funcional del módulo de interpretación clínica para múltiples patologías | |
| 4.2 | Diseño e implementación del módulo de interpretación clínica para múltiples patologías | |
| 5 | Generación automática de informes/ reportes clínicos | |
| 5.1 | Análisis de requerimientos y funcional del módulo de generación de reportes clínicos | |
| 5.2 | Diseño e implementación del módulo de generación de reportes clínicos. | |
| 6 | Produccion del software comercial “OmicsOffice(TM) for Personal Genomics” | |
| 6.1 | Versión alfa. | |
| 6.2 | Versión beta (beta testing con la FPGMX). | |
| 6.3 | Versión final commercial. |
Tabla 1
Cronograma. El estudio se realizó/ está realizando en el tiempo estimado según el cronograma a continuación:

Trabajo realizado y resultados.
A continuación se detallan los logros y resultados más relevantes alcanzados hasta la fecha:
1 Evaluación y validación del módulo de interpretación clínica.Se ha finalizado y entregado dicho módulo a los socios del proyecto, para suevaluación en un contexto clínico real. Su uso para el análisis dedatos de NGS de muestras de pacientes que sufren diversas patologías ha sido validado satisfactoriamente y mediante/ gracias a una colaboración con la unidad de genómica clínica del Hospital Ramon y Cajal.
2 Diseño de las características y funcionalidades del módulo de interpretación clínica paramúltiples muestras e inicio de la implementación de dicho modulo. Este módulo es adecuado para el análisis de múltiples patologías, tal y como se demostró en el objetivo anterior.Por lo tanto, tras un estudio exhaustivo de las necesidades actuales en el campo de la genómicaclínica, se detectó la necesidad de poder analizar múltiples muestras simultáneamente, y de formaintegrada, para el diagnóstico de ciertas enfermedades. Una vez completado el diseño del módulo deinterpretación clínica, se ha procedo a su implementación.
3 Diseño del módulo de reportes clínicos para el módulo de interpretación clínica. Se hadefinido, e implementado satisfactoriamente, el módulo de reportes clínicos para su integración con elmódulo de interpretación clínica desarrollado en el pto. 2.
4 Versión alfa 1.0 del software comercial “OmicsOffice® for Personal Genomics”. El hito másimportante en el 2013, y que engloba todos los demás, ha sido el dsesarrollo delprimer prototipo del software “OmicsOffice® for Personal Genomics”. Se trata deuna versión alfa de un primer prototipo, ya funcional, el mismo que ha podido ser enviado a los socios del proyecto parasu validación. Sobre esta base se irán añadiendo futuros desarrollos del proyecto paraculminar/ terminar en el 2014 con la versión beta final del software comercial “OmicsOffice®for Personal Genomics”.
La herramienta se ha validado hasta la fecha (2Q-2014) con el análisis de las secuencias NGS de unos 50 pacientes con patologías diferentes,incluídos en los siguientes paneles de secuenciación:
ACIDURIA D-2-HIDROXIGLUTARICA OSTEOGENESIS IMPERFECTA
ACROMATOPSIA PARAGANGLIOMA
ACROMEGALIA SINDROME DE ALSTROM
ADRENOLEUCODISTROFIA SINDROME DE BARTTER
ARTROGRIPOSIS MULTIPLE DISTAL SINDROME DE COFFIN-LOWRY
CADASIL SINDROME DE DENYS-DRASH
CAVERNOMATOSIS MULTIPLE SINDROME DE DRAVET
CHARGE SINDROME DE EHLERS-DANLOS
CORNELIA DE LANGE SINDROME DE HERMANSKY-PUDLAK
DEFICIENCIA HADHA SINDROME DE KABUKI
DEFICIT DE ARILSULFATASA-A SINDROME DE KEARNS-SAYRE
DEFICIT DE PIRUVATO DESHIDROGENASA SINDROME DE LENZ
DISPLASIA SEPTO-OPTICA SINDROME DE LESCH-NYHAN
ENCEFALOPATIA EPILEPTICA-CANALOPATIAS SINDROME DE MOWAT-WILSON
ENFERMEDAD DE LAFORA SINDROME DE PITT-HOPKINS
ESCLEROSIS LATERAL AMIOTROFICA SINDROME DE RUBINSTEIN-TAYBI
ESCLEROSIS TUBEROSA SINDROME DE SMITH-MAGENIS
EXOSTOSIS MULTIPLE SINDROME DE SOTOS
GLAUCOMA CONGENITO SINDROME DE STICKLER
GLUCOGENOSIS SINDROME DE TOWNES BROCKS
HEMIPLEGIA ALTERNANTE SINDROME DE TOWNES-BROCKS
HEMOFILIAS SINDROME DE WAARDENBURG
HIPERINSULINISMO SINDROME DE WERNER
HIPERINSULINISMO FAMILIAR SINDROME HERNS
HIPEROXALURIA PRIMARIA TELANGIECTASIA HEREDITARIA FAMILIAR
NEUROFIBROMATOSIS TRICOTIODISTROFIA
Igualmente se han analizado empleando la herramienta 12 exomas con patologías diversas:Retraso Mental, Hipoacusia neurosensorial, Distrofia muscular, Miopatía Los resultados positivos se confirmaron con secuenciación Sanger.
El módulo de interpretación clínica se ha concebido como una herramienta de análisis de secuencias de nueva generación (NGS) orientada al diagnóstico final de patologías genéticas. En el trabajo de diagnósticogenético se presenta la necesidad de interpretar los resultados de secuenciación del genoma de unindividuo (usualmente uno o varios genes del mismo) poniéndolos en correlación con el fenotipo delmismo. En este sentido la información clínica previa del paciente es tan crucial como el mismo análisis delaboratorio y debe orientar en todo momento, tanto el diseño y orientación de la analítica a realizar, comola interpretación de resultados. Por ello una herramienta de uso en este campo debe integrar todas lasfuentes de información posibles para que el genetista pueda realizar su labor.
Dada la gran heterogeneidad de casos posibles, en la práctica se dan distintos supuestos que pueden resumirse en los siguientes:
- a) Paciente con clínica que orienta a una enfermedad o síndrome concreto y conocido como enfermedad monogénica (por ejemplo Fibrosis Quística o CADASIL).
- b) Paciente con clínica que orienta a una enfermedad o síndrome conocido como enfermedad poligénica (a veces con variantes o sutipos en función de los genes concretos afectados, por ejemplo Síndrome de Noonan).
- c) Paciente con clínica que orienta a múltiples patologías genéticas distintas (por ejemplo Retraso Mental o Miopatías).
Ello se ilustra en la figura a continuación:
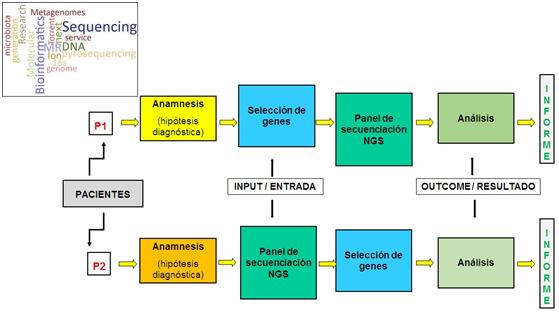
Figura 1
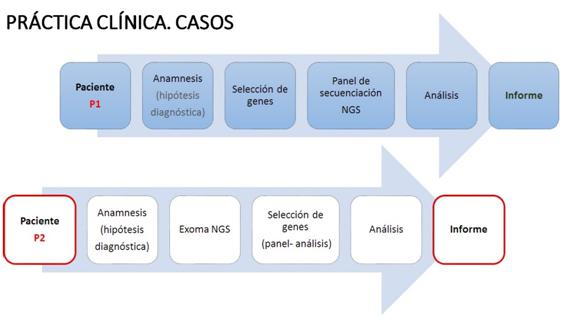 Figura 1(bis)
Figura 1(bis)
El primer y segundo casos suele comenzar con la solicitud al servicio de genética del análisis del gen o genes concretos ya conocidos. En este sentido partimos de un panel de genes pre-hecho, o panel de secuencia, para analizar en el paciente y a continuación se realiza la secuenciación correspondiente.
El tercer caso es más complejo, suele indicarse la solicitud por el fenotipo o sintomatología más importante, dejando al laboratorio de genética la selección de los genes relevantes al caso concreto en función del historial clínico. Puede realizarse la secuenciación de uno o varios genes más comunes (procedimiento común hasta ahora) o bien analizar todos los genes conocidos, quedando necesariamente abierta la posibilidad de no acertar con la selección por falta de información. En este caso actualmente es más efectivo realizar la secuenciación del exoma completo y analizar aquellos genes implicados en la clínica concreta del paciente, o panel de análisis.
En el primer caso es posible realizar una herramienta orientada a una patología única, previendo todos lasposibles variantes y circunstancias que afecta a la relación genotipo-fenotipo para dicha patología. Sinembargo esto es poco útil habida cuenta que existen muchas patologías hereditarias, todas ellas raras,salvo laboratorios especializados en una única patología lo común es recibir casos variados y de forma impredecible. Por ello es necesario un protocolo lo más sistemático posible que cubra la mayor parte de las necesidades, de modo que puedan atenderse a todos los posibles supuestos anteriormente descritos sin cambiar de herramienta.
En la FPGMX se ha evaluado la herramienta en todos los supuestos anteriores, tanto en el análisis de paneles específicos de genes, como en el análisis de exomas, que es donde se puede extraer todo su potencial.
De modo esquemático, el procedimiento estándar para el análisis de un panel de secuenciación de genes comprende los siguientes pasos:
– Carga de las anotaciones de los resultados NGS de una muestra.
– Inspección de las métricas de cobertura.
– Modificar manualmente los criterios de filtrado (opcional).
– Seleccionar la patología en GeneReviews o bien OMIM (igualmente pueden seleccionarse variantes porClinvar, o genes específicos en OMIN o RefSeq).
– Finalmente, puede extraerse un informe de resultados para incluir en el reporte/ informe, cuya apariencia es tal cual aparece en la figura 2 a continuación:
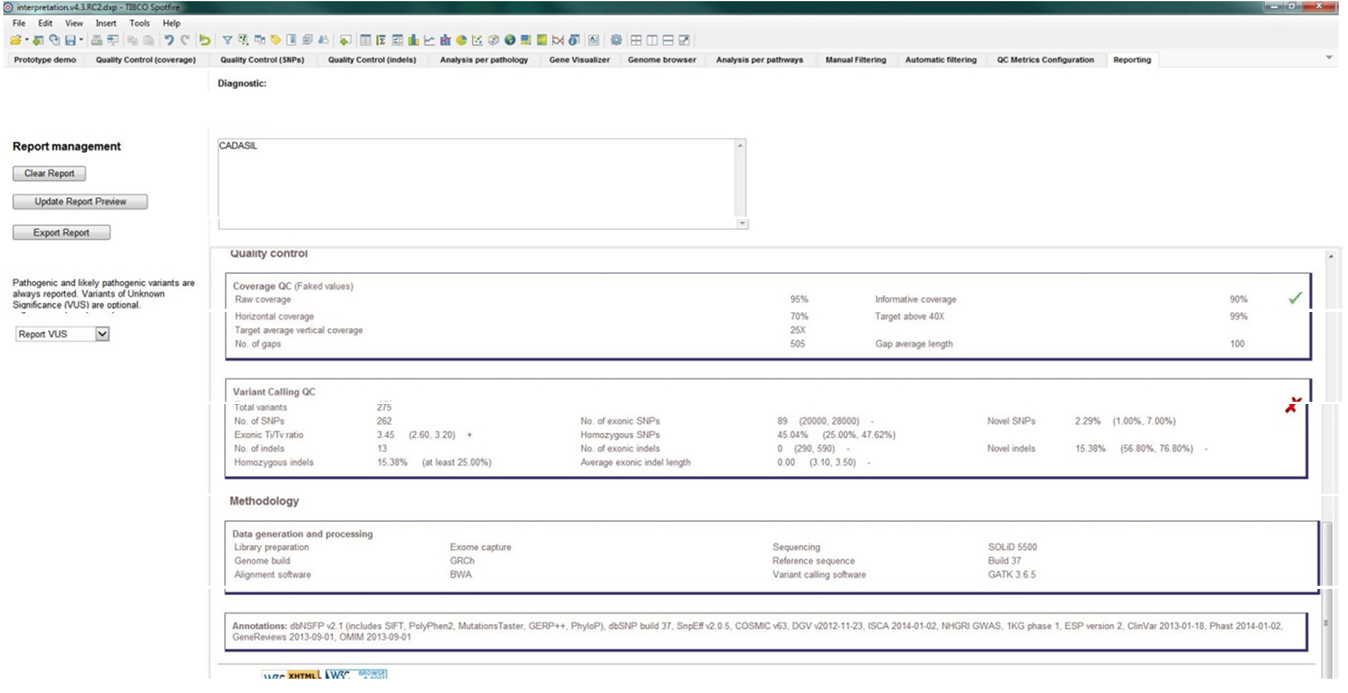
Figura 2
Casos más complejos, como es el análisis del Retraso Mental, pueden implicar la selección de variospaneles de análisis (existen muchas formas de retraso mental, con multitud de genes que en muchos casos se solapan).
Una herramienta de análisis como la del presente proyecto, requiere la capacidad deconsulta de bases de datos, tanto de acceso público como privado, que permita con un mínimo esfuerzoestar en todo momento al día en todos los supuestos clínicos que se puedan plantear.
Además de ser validado en un contexto clínico por la FPGMX, este módulo de la herramienta fue testada ademáspor un usuario temprano, la unidad de Genética Clínica del Hospital Ramon y Cajal,liderada por el Dr. Miguel Angel Moreno. Dicho usuario ha validado el prototipopara el análisis de datos genómicos de pacientes con Hipoacusias no sindrómicas.
Estatus actual. Evaluación por hitos.
El trabajo y los avances realizados en los dos últimos años son especialmente significativos y se indican, por la fase a la que pertenecen, en la tabla 2 a continuación:
| Fase | Hito: descripción de la actividad | FECHAS
(inicio – fin) |
Responsable | Completado
(%) |
|
| 3.4 | Diseño e implementación del módulo de interpretación clínica para lapatología prueba de concepto | 01/10/2012 – 30/09/2013 | FPGMX,
ITG |
¿? | |
| 3.4.3 | Evaluación del módulo de interpretación clínica. Validación del sistema | 01/10/2012 – 30/09/2013 | FPGMX,
ITG |
25/75 | |
| 4.1 | Análisis de requerimientos y funcional del módulo de interpretación clínica para múltiples patologías | 01/04 – 31/12
2013 |
FPGMX,
ITG |
33/ 67 | |
| 4.2 | Diseño e implementación del módulo de interpretación clínica para múltiples patologías | 01/10/2013 – 30/09/2014 | ITG | ¿? | |
| 5.2 | Diseño e implementación del módulo de generación de reportes clínicos (parte 1) | 01/01 – 30/06
2013 |
ITG | ¿? | |
| 6.1 | Versión alfa del software comercial “OmicsOffice ™ for Personal Genomics” (AlphaRelease v1.0) | 01/10/2013 – 30/06/2014 | ITG | ¿? | |
| … | Presentación softwarecomercial final |
Tabla 2
Además de los objetivos fijados de antemano en el proyecto, destacar que fruto de nuestra actividad en el ámbito de la genómica clínica, y de la asistencia a foros especializados para estar al día de los últimos avances del campo, hemos detectado la necesidad diseñar eimplementar un módulo para el análisis de múltiples muestras.
Ejemplos de las aplicaciones/ usos clínicos de dichos análisis múltiples son los siguientes:
Pares muestras tumor-normal.
– Desechar mutaciones germinales para poder inspeccionar las mutaciones somáticas.
– Inspeccionar mutaciones germinales conocidas que puedan predisponer al cáncer.
Caso Familiar
– Supuesto: la mutación causal es compartida dentro de las muestras afectadas.
– Duo afectados / no afectados de parientes cercanos. Maximizar mutaciones comunes, minimizarlas diferencias.
– Duo afectado / afectada de parientes lejanos. Minimizar mutaciones comunes, maximizar lasdiferencias.
– La madre y el padre no afectado y el niño afectado (u otras configuraciones).
– Otras genealogías complejas.
En el análisis de requerimientos y funcional de este módulo nos hemos centrado en los caso de uso familiares para definir losrequisitos en la interpretación de datos de NGS de múltiples muestras. El uso de cohortesno se ha considerado puesto que, de momento, no se emplea en el contexto clínico.
Se estudiaron/ estudian/ estudiaránpor lo tanto SNPs e indeles asociados apatologías monogénicas en los diseños experimentales conocidos. El diseño del módulo también permitegeneralizarlo para el uso en el análisis de pares de muestras tumor-normal y, al igual que el módulo de interpretación clínica, no tendrá ninguna limitación encuanto al tipo de patología.
Finalmente, queremos destacar que el desarrollo de la versión alfa 1.0 del software, para la interpretación clínica de NGS, se completó en el 2014. Si bien no se han implementado completamente las funcionalidades definidas para el software, por tratarse de una versión alfa, y lasimplementadas pueden ser modificadas, eliminadas o complementadas por otras nuevas, esta versión cumple su misión como prueba de la aplicación en el trabajo cotidiano de laFPGMX para el análisis de muestras individuales. Las funcionalidades de carga automática de datos y decomprobación de la calidad del análisis es de gran importancia para este fin, de lo contrario no será utilizado para casos de rutina.
Esta versión alfa es el resultado de varias iteraciones de prototipos, proceso que comprende (i) definición de requisitos, (ii) creación de prototipos y evaluación, tanto por los expertos clínicos (esto es la FPGMX y la Unidad de Genética Clínica del Hospital Ramón y Cajal) como internamente enIntegromics.
El flujo de trabajo realizado para el desarrollo de la versión alfa 1.0 del software se muestra en la figura 3 a continuación:
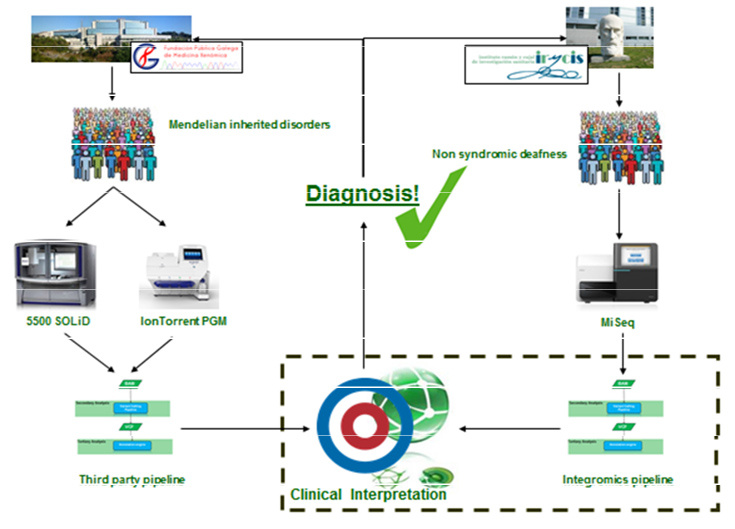
Figura 3. Flujo de los datos en ApliClinics
La aplicación se ha implementado en la plataforma TibcoSpotfire, una solución de ‘businessinteligence’ que permite la configuración de los cuadros de mando personalizados. En este caso se empleó la interfazgráfica de Spotfire para desarrollar los cuadros de mando de interés. La interfaz de programación deaplicaciones Spotfire avanzada se ha usado para las interacciones de uso más complejos tales como carga automática de datos y presentación de informes de funcionalidades. Otrainformación clínicamente relevante se integrómediante implementaciones personalizadas.
Estatus actual.
Proyectofinalizado el dd/mm/2014
Resultados y publicaciones asociados
ND
Alineamiento actual/ real del proyecto/ estudio con las estrategias gubernamentales para la promoción de la innovación
En líneas generales, la contribución del proyecto a cada uno de los cinco ejes de actuación que conforman el llamado «Pentágono de la Innovación» ha sido:
1 En cuanto a la creación de un entorno financiero favorable a la innovación empresarial: el socio empresarial en este consorcio, Integromics, ha recibido el impulso necesario para realizar un nuevo proceso de innovación empresarial, consistente en, partiendo de su experiencia en bioinformática en ciencias de la vida, adentrarse en el área de Medicina Personalizada. Esta innovación se implementará en un nuevo producto de software, que incluirá funcionalidades para analizar datos masivos de re-secuenciación genómica para las aplicaciones clínicas, incluyendo el diagnóstico. Para las tecnologías de genómica más recientes, también conocidas comoNextGenerationSequencing (NGS), la interpretación de los datos representa/ es reconocida como un obstáculo importante en la adopción de la medicina personalizada, en la asistencia sanitaria ordinaria,debido a su alto coste y tiempo de análisis requerido. Esta innovadora solución/ herramienta bioinformática, llamadaOmicsOffice®, acelerará/ hará posible/ contribuirá decisivamente ala adopción de las últimas técnicas de NGS para el análisis de datos genómicos en el ámbito asistencial ordinario, propiciando una disminución de la carga de trabajo de los facultativos y aumentando la eficiencia del SNS.
2 La dinamización de los mercados innovadores mediante la regulación y la compra pública: se está creando un mercado innovador, el mercado de las aplicaciones bioinformáticas para el análisis genómico en el ámbito clínico ordinario.
3 El fortalecimiento de la cooperación territorial en materia de promoción de la innovación: las 2 entidades asociadas pertenecen a regiones diferentes de España, la CCAA Galicia (FPGMX) y Andalucía (ITG); Integromics además tiene otra sede en la CCAA de Madrid.
4 La proyección internacional de las actividades innovadoras: gracias a este proyecto se ha empezado una línea de innovación a través de actividades de I+D que sin duda se alimentará de colaboraciones a nivel internacional mediante futuros proyectos y alianzas estratégicas con líderes en este campo. Así los avances y primeros resultados del proyecto se presentaron en el congreso “ClinicalGenomics&InformaticsEurope 2013” organizado por Cambridge HealthTech&Bio-ITWorld . Así mismo, un socio de Integromics, PerkinElmer,ha mostrado un gran interés en colaborar y/o llevar a cabo desarrollos conjuntos.
5 El capital humano mediante la promoción de la formación y la provisión de talento investigador e innovador al sector empresarial: el presente proyecto presenta la oportunidad de crear tejido público-privado en investigación e innovación en bioinformática clínica, abriendo nuevas posibilidades en sectores como la medicina personalizada, de evidente proyección estratégica hacia el futuro de nuestro modelo productivo. Así el personal implicado en este proyecto, gracias a la ayuda recibida, podrá ser el núcleo de nuevos equipos de profesionales altamente cualificados que, a su vez, serán nuevas oportunidades de empleo de calidad en España.





